O processo evolucionário como um todo pode ser descrito como a busca de uma espécie pela maximização da propagação de genes ao longo do tempo, ou seja, obter a linhagem mais longa possível de descendentes com as mesmas características genéticas fundamentais por meio de alterações genéticas hereditárias secundárias que produzam mudanças fenotípicas entre as gerações que aumentem as chances de sobrevivência e reprodução. Por definição, dado que as múltiplas pressões e desafios do ambiente tendem a mudar de forma significativa com o tempo, as espécies que não evoluem tendem a se extinguir [1].
Em se tratando de patógenos virais como o SAR-CoV-2, as pressões e desafios envolvem não apenas elementos como a imunidade natural, eventual imunidade de rebanho e o desenvolvimento e uso de antivirais, mas também o acesso a novos indivíduos para infectar. Nesse contexto, evoluir implica em simultaneamente aumentar a taxa de transmissão e diminuir a letalidade de modo a se maximizar a disseminação e permanência numa população [2].
É importante observar que o processo evolucionário é essencialmente estocástico, dependendo de acasos envolvendo tanto o ambiente quanto a ocorrência de mutações genéticas. No que se refere ao SARS-CoV-2, isso significa que a sucessão temporal de variantes não ocorre necessariamente por meio de passos unidirecionais orientados para uma redução da transmissibilidade e letalidade, podendo pontualmente haver aumento em uma ou ambas as coisas entre uma variante específica e a seguinte. Isso tem levado alguns a deduzir que não se pode ter certeza de que novas versões mais perigosas venham a emergir [3][4]. Contudo, embora a incerteza efetivamente exista a cada instante, a convergência probabilística é matematicamente inexorável, de modo que, uma sequência de variantes ao longo do tempo tenderá de fato a um máximo de transmissibilidade e a um mínimo de letalidade ainda que oscilações aleatórias possam surgir na trajetória.
A sucessão de variantes do SARS-CoV-2 no Brasil pode ser acompanhada pelo Dashboard Rede Genômica da Fiocruz [5], o qual usa dados genômicos do sistema GISAID para construir curvas de prevalência como a apresentada na Figura 1.
Ao longo do tempo, constatou-se a existência de mais de uma dúzia de variantes de interesse do SARS-CoV-2 no Brasil, com a Omicron emergindo e tornando-se dominante com rapidez sem precedente em janeiro de 2022, iniciando uma sucessão de subvariantes, cada uma com aumento igualmente rápido da sua prevalência em relação à anterior.
Combinando-se os dados do Sistema Fiocruz [5] acerca da prevalência das variantes do SARS-CoV-2 e do SRAG do OpenDataSUS [6] sobre os casos e mortes atribuídos à Covid-19 segundo a data de ocorrência, tem-se o que é mostrado na Figura 2.
Observa-se claramente que a sequência de prevalências de variantes do SARS-CoV-2 no país se faz acompanhar de variações substanciais na trajetória de casos e de óbitos atribuíveis à Covid-19.
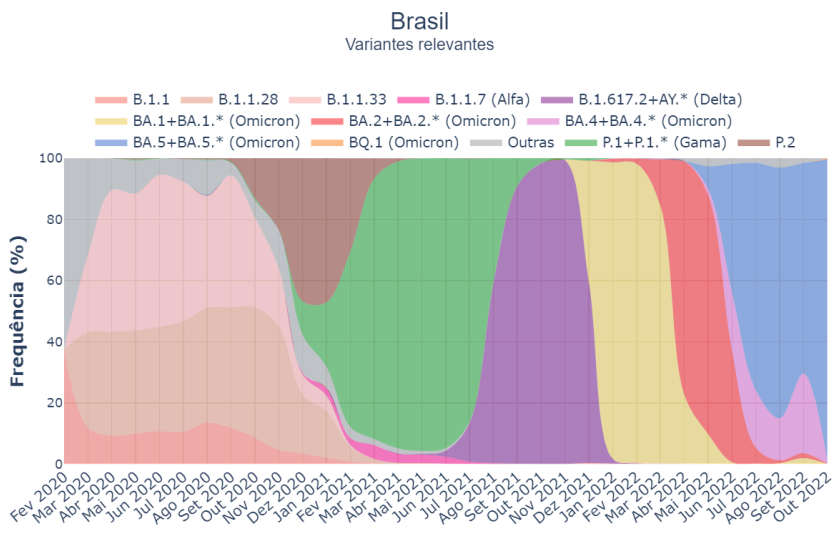
Fonte: Rede Genômica da Fiocruz [5].
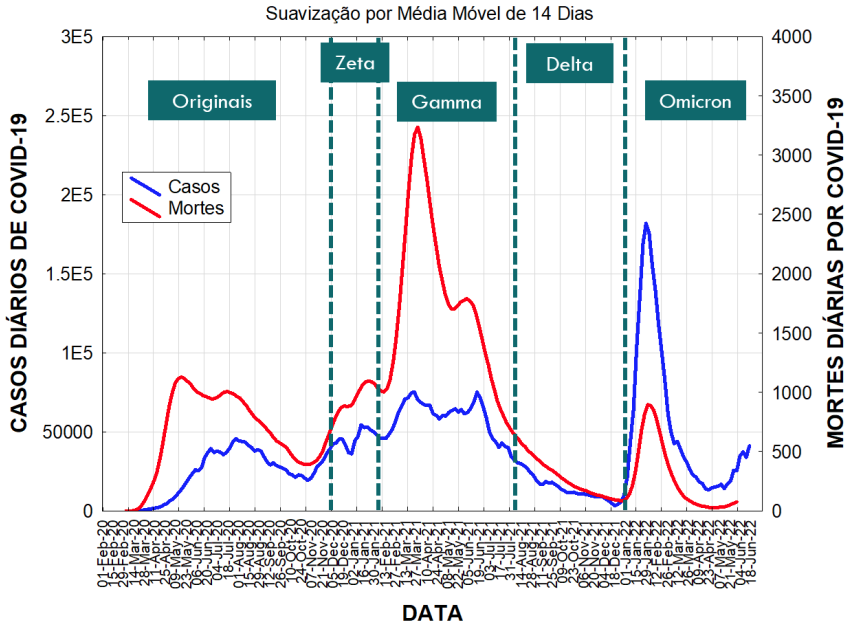
variante prevalente.
Por exemplo, quando predominavam as variantes originais (principalmente B.1.1, B.1.1.28 e B.1.1.33) teve-se um grande aumento nos óbitos e relativamente pouco nos casos, possivelmente refletindo limitações iniciais no diagnóstico da Covid-19. Já durante o domínio da Gamma (P.1+P.1.*) houve grande aumento nos óbitos mas relativamente pouco nos casos. Mais adiante, veio a Omicron (inicialmente com a BA.1+BA.1.* e a BA.2+BA.2.*), com grande elevação nos casos mas pouca nos óbitos, indicando grande transmissibilidade e baixíssima mortalidade.
Os dados do Sistema Fiocruz [5] apontam que as subvariantes iniciais da Omicron, ou seja, BA.1+BA.1.* e a BA.2+BA.2.*, foram sucedidas pela BA.4+BA.4.* e, principalmente, a BA.5+BA.5.*, com alguns indícios iniciais da BQ.1. Contudo, segundo os dados dos atestados de óbito dos cartórios nacionais [7], de longe os mais precisos e atualizados, isso não se fez acompanhar de aumento na quantidade de óbitos ao longo do tempo, a qual continua a cair.
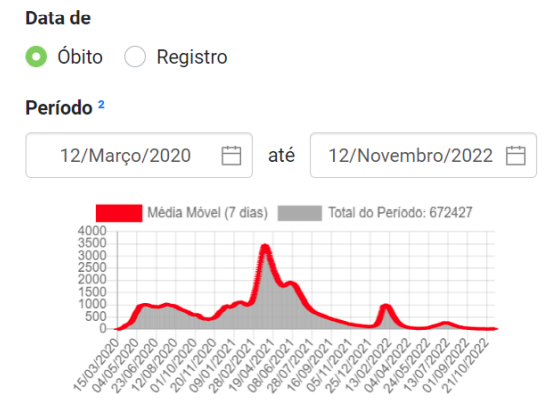
Central de Informações do Registro Civil – CRC Nacional.
Dado o exposto, é forçoso concluir que os recentes relatos anedóticos de elevação na quantidade de casos de Covid-19 em certas partes do país, porém com mortalidade ainda em queda, são consistentes com o SARS-CoV-2 aproximando-se do seu ponto de ótimo evolucionário de máxima transmissibilidade e mínima letalidade. Considerando-se que existe uma inegável e forte associação entre a gravidade típica dos casos e a mortalidade, a constatação de aumento na positividade de testes e do número de internações parece refletir mais uma grande preocupação e cautela com a doença do que sua efetiva severidade. Conclui-se, portanto, não haver motivo para a preocupação com uma eventual “onda” adicional da Covid-19 no Brasil, inexistindo necessidade de se adotar medidas extraordinárias.
Referências
[1] Hall, Brian K.; Hallgrímsson, Benedikt (2008). Strickberger’s Evolution (4th ed.). Sudbury, Massachusetts: Jones and Bartlett Publishers. ISBN 978-0-7637-0066-9. LCCN 2007008981. OCLC 85814089.
[2] Geoghegan JL, Senior AM, Di Giallonardo F, Holmes EC. Virological factors that increase the transmissibility of emerging human viruses. Proc Natl Acad Sci U S A. 2016 Apr 12;113(15):4170- 5. doi: 10.1073/pnas.1521582113. Epub 2016 Mar 21. PMID: 27001840; PMCID: PMC4839412.
[3] Holmes, Bob. How Viruses Evolve Pathogens that switch to a new host species have some adapting to do. How does that affect the course of a pandemic like COVID-19? Knowable magazine, July 17, 2020
[4] Eva Botkin-Owaci. Do Viruses Such as the Coronavirus Become Less Deadly? Understanding how viruses evolve can help us understand what future variants of the coronavirus we might see—and whether or not we should. News@Northeastern. In the Inetrnet at: https://news.northeastern.edu/2021/12/13/virus-evolution/
[5] FIOCRUZ. Dashboard Rede Genômica – Enabled by data from GISAID. Vigilância Genômica do SARS-CoV-2 no Brasil. Fundação Oswaldo Cruz – FIOCRUZ, Ministério da Saúde. na Internet em: http://www.genomahcov.fiocruz.br/dashboard/
[6] OpenDataSUS. SRAG 2020/2021 – Banco de Dados de Síndrome Respiratória Aguda Grave – incluindo dados da COVID-19. Ministério da Saúde, Governo Federal. Na Internet em: https://opendatasus.saude.gov.br/dataset/bd-srag-2020
[7] Portal da Transparência – Painél Registral – Especial Covid-19. Na Internet em: https://transparencia.registrocivil.org.br/especial-covid


